深度学习如何进行MRI图像右心室分割
人类心脏是一台令人惊叹的机器,它能持续运转长达一个世纪而不失灵。测量心脏功能的关键方法之一是计算其射血分数,即每搏输出量占心室舒张末期容积量的百分比。而测量这个指标的第一步依赖于对心脏图像心室的分割。
问题描述
开发一个能够对心脏磁共振成像(MRI)数据集图像中的右心室自动分割的系统。到目前为止,这主要是通过经典的图像处理方法来处理的。而现代深度学习技术有可能提供更可靠、更自动化的解决方案。
2016年由Kaggle赞助的左心室分割挑战赛中的三名获奖者都采用了深度学习解决方案。然而,分割右心室(RV)则更具挑战性,因为:
在腔内存在信号强度类似于心肌的小梁; RV复杂的新月形;分割根尖图像切片的难度;个体之间的室内形状和强度存在相当大的差异,特别是在不同的疾病病例之间。
撇开医学术语不谈,要识别RV就更困难了。左心室是一个厚壁圆环,而右心室是一个形状不规则的物体,有薄的壁,有时会与周围的组织混合在一起。这是MRI快照右心室内壁和外壁(心内膜和心外膜)的手工绘制轮廓:

这是一个分割起来很容易的例子。这一个比较困难:

而这对于没有经过训练的眼睛来说完全是一个挑战:

事实上,与左心室相比,医生需要耗费两倍的时间来确定右心室的体积并生成结果。这项工作的目的是建立一个高度准确的右心室自动分割深度学习模型。模型的输出是*分割掩码*,即一个逐像素的掩码,用来表示某个像素是否是右心室的一部分或只是背景。
数据集
对于当前这个问题,深度学习需要面对的最大挑战是数据集太小。 数据集(可以访问这里)仅包含了来自于16例患者的243张医师分割的MRI图像。 另外还有3697张未标记的图像,这对于无监督或半监督技术可能会有用,但是我将这些放在了一边,因为这是一个监督学习问题。 图像尺寸为216&TImes;256像素。
由于数据集过小,人们可能会怀疑无法将其一般化到尚未看到的图像!但是很不幸,医疗上标记过的数据非常昂贵,并且很难获取到。对数据集进行处理的标准程序是对图像应用仿射变换:随机旋转、平移、缩放和剪切。此外,我实现了弹性变形,也就是对图像的局部区域进行拉伸和压缩。

应用这种图像增强算法的目的是为了防止神经网络只记住训练的样例,并强迫其学习RV是一个实心的、月牙形的、方向任意的物体。在我实现的训练框架中,我会随时对数据集应用图像变换算法,这样,神经网络就会在每次训练时看到新的随机变换。
由于大多数像素都属于背景,所以各个种类之间分布不平衡。如果将像素强度归一化在0和1之间,那么在整个数据集中,只有5%的像素属于RV腔的一部分。
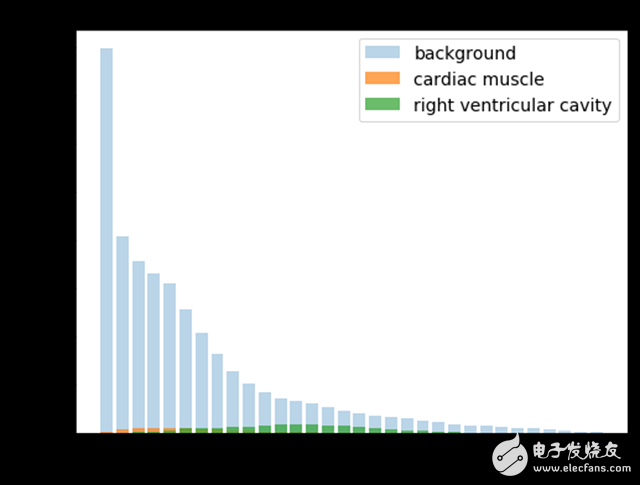
在创建损失函数时,我尝试了重加权方案来平衡种类的分布情况,但最终发现未加权平均算法的表现最好。
在训练中,有20%的图像被取出来作为验证集使用。 RV分割挑战赛的组织者有一个单独的测试集,它由另外32个患者的514张MRI图像组成。我提交的预测轮廓就是使用这个测试集进行最终评估的。
还需要有一种方法来对数据集上的模型性能进行量化。RV分割挑战赛的组织者选择使用了戴斯系数。模型会输出一个掩码*X*来描述RV,而戴斯系数将*X*与由医师创建的掩码*Y*通过以下方式进行比较:

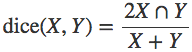
计算值是交叉区域与两区域之和的比率的两倍。对于不相交的区域,值为0;如果两区域完全一致,则值为1。
下面我们来看下模型架构。
U-net:基线模型
由于我们只有四周的时间来完成这个项目,所以我想尽快实现一个基线模型并让它运行起来。我选择了由Ronneberger、Fischer和Brox提出的u-net模型,因为它曾在生物医学分割项目中取得过成功,而且它的作者能够通过使用积极的图像增强和逐像重新加权算法并仅基于*30张图片*来训练网络。
u-net架构由一个收缩路径组成,就是将图像折叠成一组高级特征,随后是使用特征信息构建像素分割掩码的扩展路径。 u-net独特的地方就是它的"复制和合并"连接,这些连接能够将信息从早期特征图传递到构建分割掩码网络的后续部分。作者指出,这些连接允许网络同时并入高级特征和像素方面的细节。
我们使用的架构如下所示:
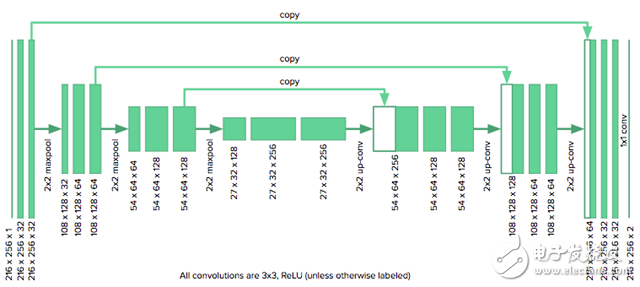
由于我们图像尺寸是u-net作者原本考虑的一半大小,所以我们需要将原始模型中的降采样层数从4个减少到3个来适应网格。我们也要用零来填充卷积,以保持图像的大小不变。该模型是用Keras实现的。
如果没有图像增强,u-net在训练数据集上的戴斯系数能达到0.99(0.01),这意味着该模型具有足够的能力来捕获RV分
- 机器学习算法盘点:人工神经网络、深度学习(07-02)
- 2016年人工智能与深度学习领域的十大收购(07-26)
- AI/机器学习/深度学习三者的区别是什么?(09-10)
- 深度学习的硬件架构解析(10-18)
- 麻省理工科技评论评选的14大医疗领域突破科技(上)(10-14)
- 探秘机器人是如何进行深度学习的(09-18)
