應用系列之一 - 生醫資訊之BLAST演算法
时间:10-02
整理:3721RD
点击:
前言:
本篇應用基於基因Alignment演算法做一個實際的編寫,BLAST(1990年)是其中一個相當熱門的演算法,因此就以自己理解的方式來編寫一個小型Demo,官方(NCBI)https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastHome已經提供完整的BLAST工具可作測試使用,此Demo主要反映BLAST的演算法邏輯
準備:
1. C6655開發板
2. TL-XDS200模擬器,箱內有附
3. 12V 2A Adapter(電源),箱內有附
4. USB TypeB傳輸線,箱內有附
1. 問題描述:給定數個已知的基因序列資料庫,假設我們要分辨一個未知物種和已知物種的差異性或者帶有特定的功能基因序列,我們擁有該物種一個基因序列片段,我們如何在這資料庫中去比對最有可能相符合的基因序列來決定該物種的資訊,因為資料相當龐大(~上TB),因此BLAST就發展用來解決這個問題
2. 基本流程和步驟如下說明:
- Step1: 利用Hash Table將資料庫的基因序列做小片段裁切,本應用假設3個基因組為一個單位
- Step2: 給定一個特定的Threshold值,將待測的序列做初步篩選
- Step3: 篩選方式即使用Hash Table來做映射,因一單位為3,所以至少有完全符合配對的3個基因組會被篩選出來
- Step4: 篩選完後,即代表一個Hit,每一個Hit的序列兩邊繼續延伸,當完全符合時+5分,不符合的配對-4分,利用DP Table來實作
- Step5: 從待測的序列配對出來總分數較高的為一個MSP,當兩端延伸低於一個分數X時候停止延伸
- Step6: 在眾多的MSP輸出分數最高的基因序列配對
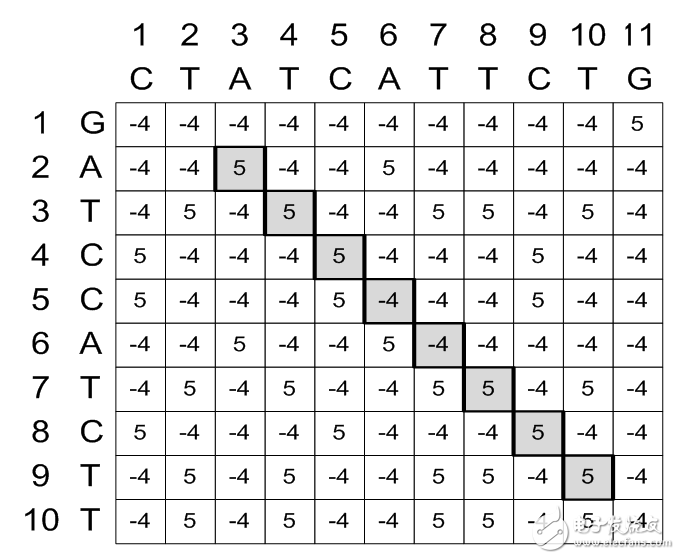
圖為Alignment Matrix找出Maximal Segment Pair(MSP)
3. 本篇應用初始設定值如下,這些值將會影響結果,資料庫序列和待測序列為預先分別給定:
- 最低分數 X = 4
- Threshold值 T is 3-mers (hits) = 5x3 = 15
- 資料庫序列長度 Database sequence length = 140
- 待測的序列長度 Segment sequence length = 7
- 符合配對 Identities: +5
- 不符合配對 Mismatches: -4
- One Task in SYS/BIOS
- A = 0
- C = 1
- G = 2
- T = 3
4. 將開發板連接模擬器,再用USB TypeB傳輸線連上電腦後,打開CCS IDE,創建SYS/BIOS專案,這裡可以參考創龍的文檔,這裡將不再贅述
5. 打開專案工程中main.c檔案,輸入以下程式碼:
- /*
- * ======== main.c ========
- */
- #include
- #include
- #include
- #include
- #include
- const int X=4;
- const int length=140;
- const int entry=64;
- int database[]={
- 0,1,3,2,1,2,3,
- 3,3,2,2,1,0,3,
- 2,1,3,0,1,2,1,
- 3,2,1,2,3,1,2,
- 3,1,2,3,1,2,3,
- 1,3,2,3,1,2,3,
- 2,2,3,2,3,1,2,
- 2,1,2,3,1,2,1,
- 2,2,1,2,3,3,1,
- 3,2,3,1,3,2,1,
- 2,1,2,3,2,0,1,
- 0,2,2,1,0,2,3,
- 3,2,1,2,2,3,1,
- 2,2,0,3,0,0,2,
- 3,2,1,2,3,1,0,
- 2,3,1,2,1,1,2,
- 2,1,1,3,3,2,0,
- 3,1,1,3,2,1,2,
- 3,2,1,1,3,2,3,
- 0,2,3,2,2,2,1
- };
- int seg[]={0,1,2,3,2,1,0};
- int hash[64][141];
- int dp[140][141];
- /*struct listNode
- {
- int a;
- listNode *nl;
- };*/
- void table(int data[])
- {
- int i,j;
- for(i=0;i 0)
- {
- for(j=1;j temp)
- temp=dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0];
- }
- else
- {
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]+3+k]=-4;
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0]-=4;
- if(dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0] -1)
- {
- if(data[i+l]==database[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]+l])
- {
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]+1+l]=5;
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0]+=5;
- if(dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0]>temp)
- temp=dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0];
- }
- else
- {
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]+1+l]=-4;
- dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0]-=4;
- if(dp[hash[data[i]*16+data[i+1]*4+data[i+2]*1][j]][0] 0)
- {
- System_printf("hash=%d\n",i);
- for(j=0;j max)
- max=dp[i][0];
- }
- System_printf("\n");
- }
- System_printf("Result of the Alignment\n");
- for(i=0;i =max)
- System_printf("Maximum score=%d,started in index=%d\n",dp[i][0],i);
- }
- System_printf("exit taskFxn()\n");
- }
- /*
- * ======== main ========
- */
- Int main()
- {
- Task_Handle task;
- Error_Block eb;
- System_printf("enter main()\n");
- Error_init(&eb);
- task = Task_create(taskFxn, NULL, &eb);
- if (task == NULL) {
- System_printf("Task_create() failed!\n");
- BIOS_exit(0);
- }
- BIOS_start(); /* does not return */
- return(0);
- }
6. 其中Hash Table和DP Table的資料格式如下:

上圖為Hash Table,第一個index用來記錄映射到該3-mers的index總數,之後記錄起始映射到的資料庫index
下圖為DP Table,第一個index用來記錄該Hit index起始位置為延伸的總分,之後紀錄符合和不符合的加減分
7. 打開專案的app.cfg檔案,設定BIOS的Heapsize和輸出用的bufsize,如下圖
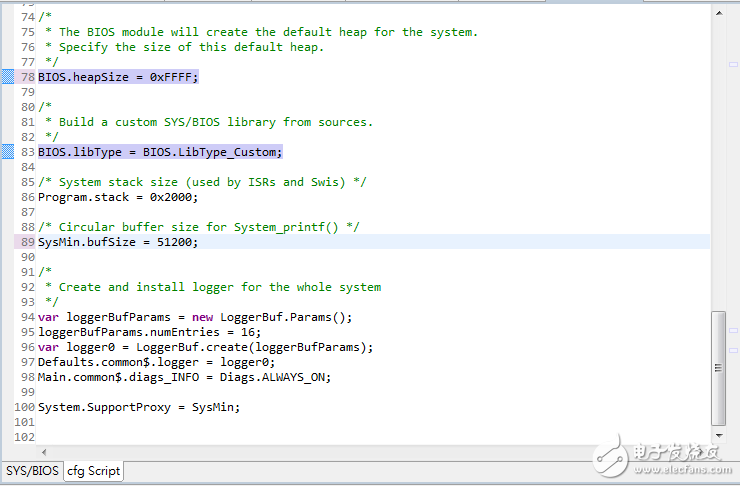
設定BIOS參數
- BIOS.heapSize = 0xFFFF;
- SysMin.bufSize = 51200;
8. 將GEL組態設定檔連結好,並啟用,之後在專案上按下滑鼠右鍵,選擇Build Project後,無錯誤的話點擊滑鼠右鍵選擇開啟Debug Session
9. 按一下Resume按鈕,運行一下模擬器上掛載好的程式,等待片刻後,按下Suspend按鈕暫停程式
10. 在ROV視窗中選擇SysMin > OutputBuffer頁籤
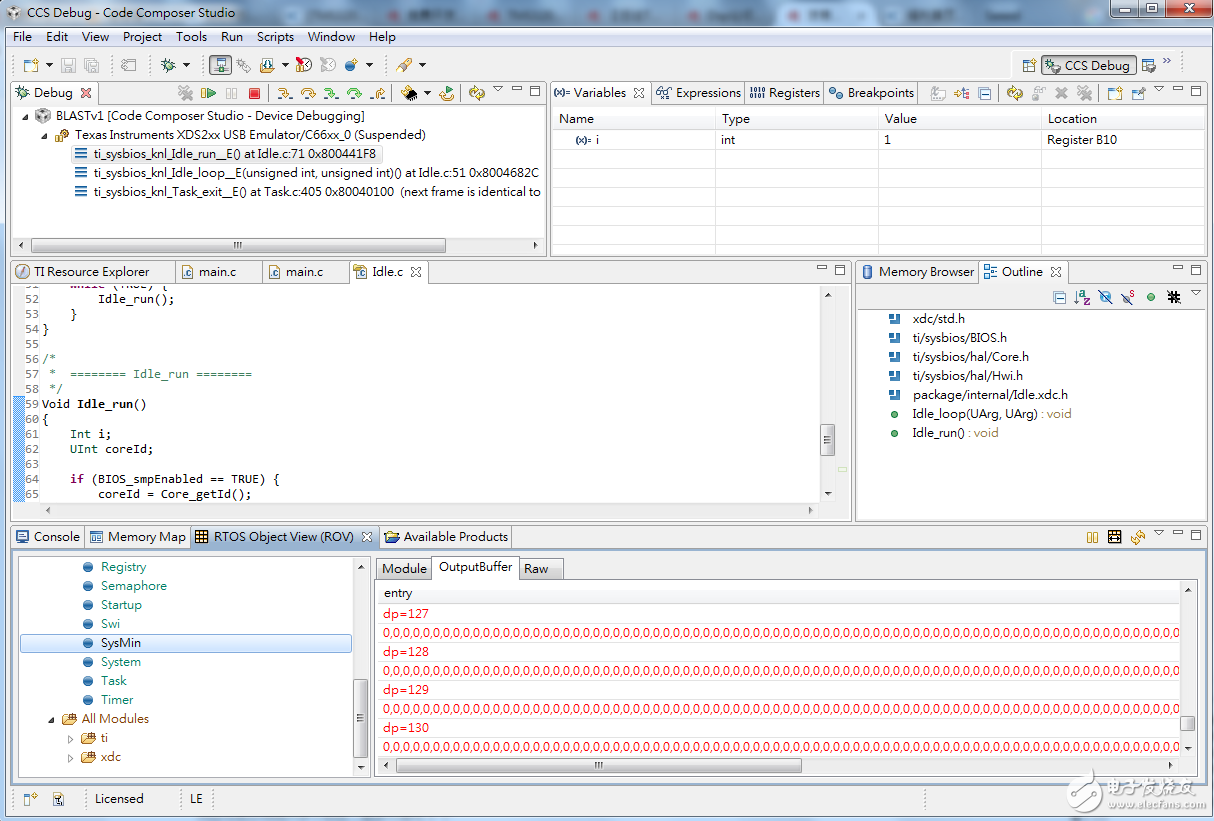
選擇OutputBuffer觀看輸出訊息
11. 按一下右上角的放大視窗,將輸出訊息拉到最底下,可觀看完整的輸出結果
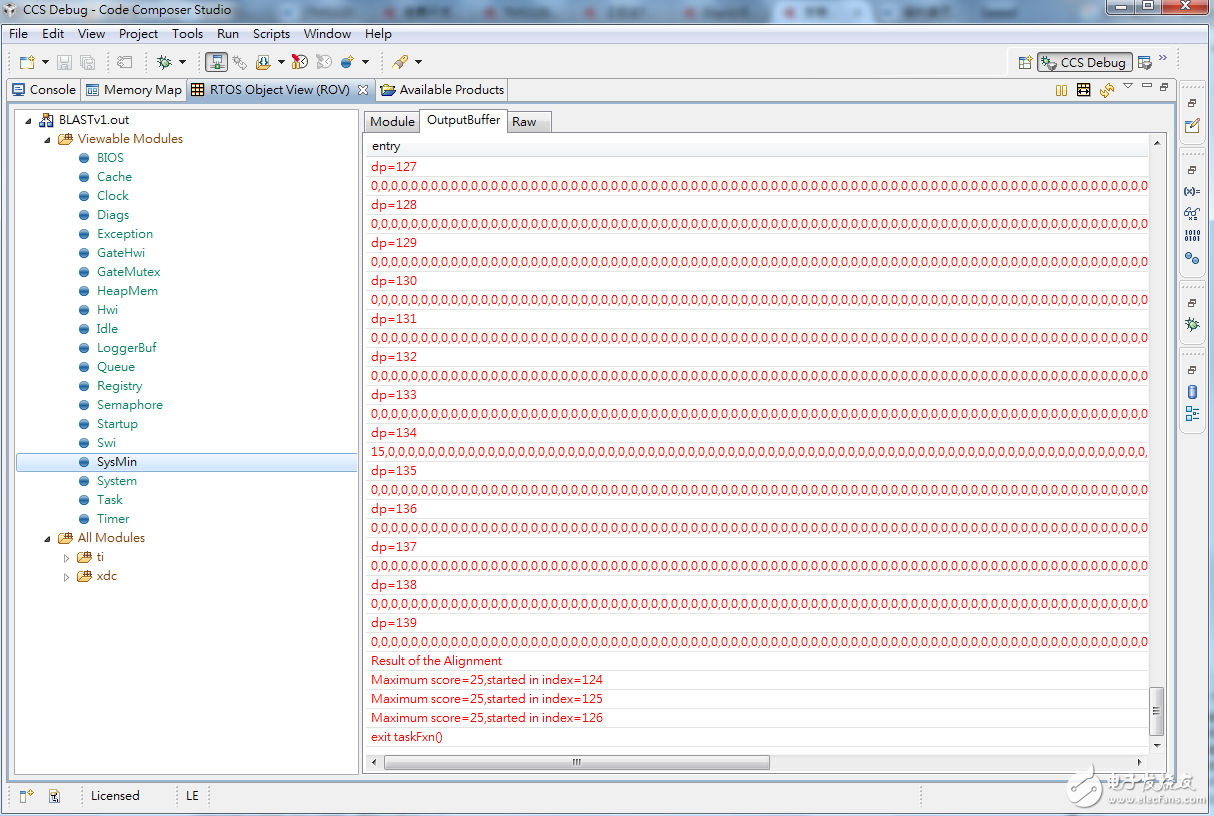
資料庫序列中index為124,125和126的三個位置有最大MSP
12. 接下來我們來驗證演算法Demo是否正確,追溯Demo第124,125和126位置的配對,其實因為3-mers相鄰都符合,所以這3行的DP Table是一樣的,如下圖

圖中央分別是124,125和126
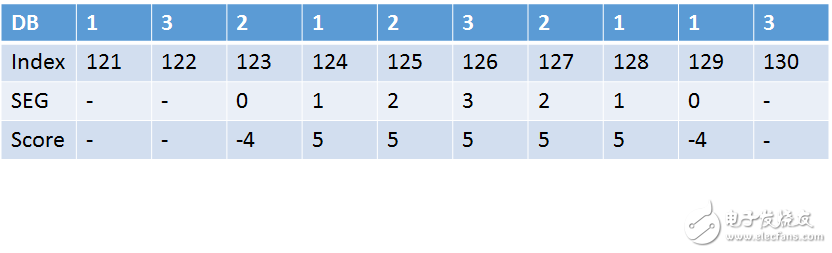
對照OutputBuffer的輸出訊息,總分取5+5+5+5+5=25分,因此驗證成功!
小結:
BLAST在生醫資訊領域中DNA的alignment是經典的演算法之一,透過大型Server可加快Alignment的速度,並推廣至大數據的DNA資料,透過這個簡易的Demo來體會BLAST演算法的精隨,本文實際應用DSP和SYS/BIOS系統來做一個實際的演算法,利用RTOS的特性,可加快處理速度將結果輸出,並如果將程式分成N個Task,將有效縮短本Demo的執行時間,供小伙伴們參考,下篇將提出另一個在alignment之前的correction高效演算法
